品牌:金开瑞
起订:11
供应:10001
发货:3天内
信息标签:全方位表观遗传调控研究技术—金开瑞生物,提供服务,商务服务,其他未分类


一、表观遗传学简介
表观遗传学是研究基因的核苷酸序列不发生改变的情况下,基因表达的可遗传的变化的一门遗传学分支学科。DNA甲基化(DNAmethylation)、组蛋白修饰(histone modification)、基因组印记(genomic imprinting)、RNA编辑(RNA edi-ting)、基因沉默、核仁显性、休眠转录子激活和性别相关性基因剂量补偿效应等都是典型的表观遗传现象。
表观遗传调控研究技术分为转录前调控研究技术和转录后调控研究技术。转录前调控研究技术主要包括:MSP、BSP、ChIP、EMSA、DNA Pull down、酵母单杂交(Y1H);转录后调控研究技术主要包括RIP、RNA Pull down 、双荧光素酶报告系统等。
金开瑞致力于ChIP、EMSA、DNA/RNA pulldown、RIP、双萤光素酶系统、MSP(甲基化特异性PCR法)、BSP(亚硫酸氢钠处理后测序法)、MeDIP-seq等实验技术服务。除了提供**的技术服务外,我们还可以为您提供表观遗传学整体项目设计,整体项目实施,整体项目跟踪服务,金开瑞多年的实验和项目管理经验,将助力您表观遗传研究。
整体研究框架
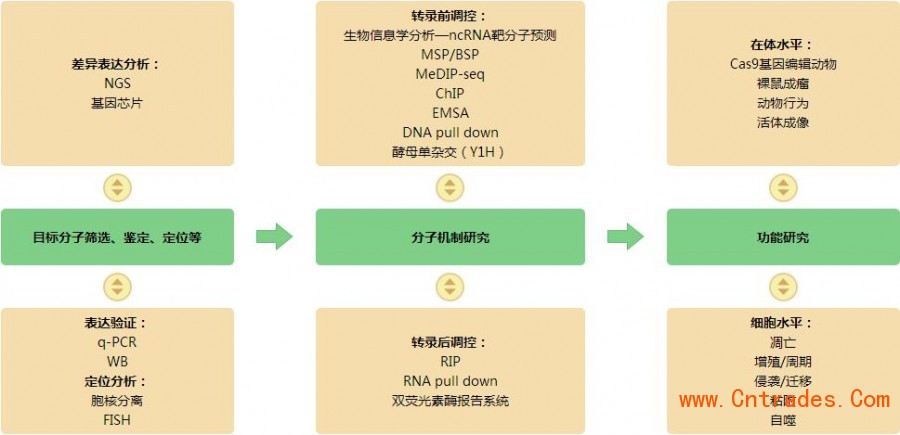
二、转录前调控研究技术
MSP(甲基化特异性PCR)
甲基化特异性是指用亚硫酸氢钠处理基因组DNA,未甲基化的胞嘧啶变成尿嘧啶,而甲基化的胞嘧啶不变,然后用3对特异性的引物对所测基因的同一核苷酸序列进行扩增。扩增产物用DNA琼脂糖凝胶电泳,凝胶扫描观察分析结果。
技术流程
提取基因组DNA,并定量—亚硫酸氢钠处理DNA—DNA修饰纯化后回收—引物设计合成,PCR检测—琼脂糖凝胶电泳
技术运用
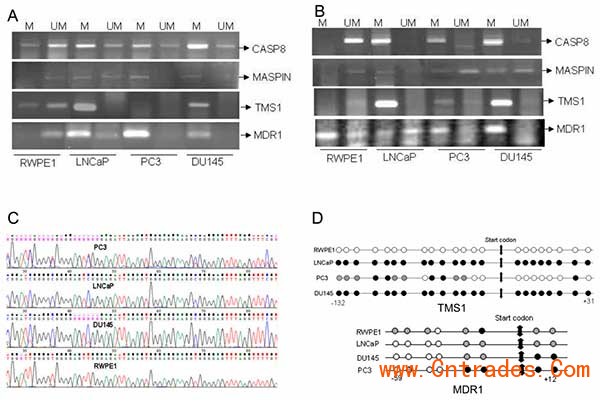
(A): Methylation specific PCR of CASP8, MASPIN, TMS1 and MDR1 in prostate cancer cells. DNA from prostate cancer cells were Bisulfite modified using EZ methylation kit (Zymo Research) and then PCR done using methylation specific primers and unmethylation specific primers. Because in the cells sometimes methylated and unmethylated both copies are present, so depending upon the number copies they get amplified. (B): Methylation specific PCR of CASP8, MASPIN, TMS1 and MDR1 in 5-aza-dC (5µM/48hrs) treated prostate cancer cells. (C) Bisulfite sequencing chromatogram of TMS1 gene showing methylation in LNCaP, DU145 and PC3, partial methylation in PC3 and Unmethylation in RWPE1. (B): Diagrammatic representation of methylation pattern of TMS1 and MDR1 gene at different promoter positions in RWPE1, LNCaP, DU145 and PC3. Dark circles (●) represents methylated sites, shadowed circles (An external file that holds a picture, illustration, etc.Object name is nihms159165ig1.jpg) represent partially methylated sites and blank circles (○) represents unmethylated sites in TMS1 and MDR1 promoter region.
(Mishra DK, Chen Z, Wu Y, Sarkissyan M, Koeffler HP, Vadgama JV. Global methylation pattern of genes in androgen-sensitive and androgen-independent prostate cancer cells. Mol Cancer Ther. 2010 Jan;9(1):33-45.)
亚硫酸氢盐处理后测序法(bisulfite sequencing PCR BSP)
亚硫酸氢盐处理后测序法:这种方法一度被认为是DNA甲基化分析的金标准。它的过程如下:经过亚硫酸氢盐处理后,用PCR扩增目的片段,并对PCR产物进行测序,将序列与未经处理的序列进行比较,判断CpG位点是否发生甲基化。这种方法可靠,且**度高,能明确目的片段中每一个CpG位点的甲基化状态,但需要大量的克隆测序,过程较为繁琐、 昂贵。
技术流程
基因组DNA的提取—亚硫酸氢钠修饰基因组DNA—引物设计及修饰后DNA用于PCR—PCR产物的凝胶回收—PCR产物与T载体的连接和转化、克隆筛选
技术运用
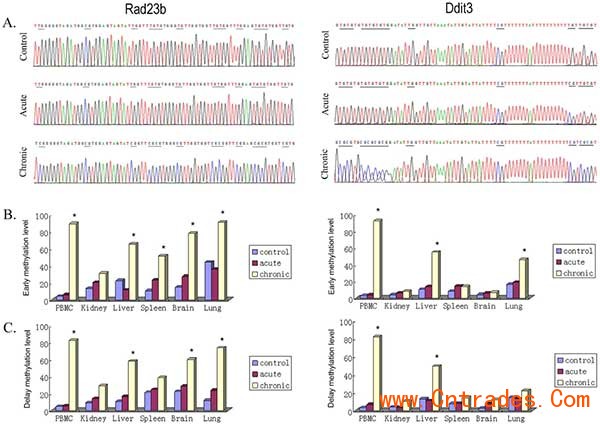
Rad23b and Ddit3 methylation determined by BSP. Bisulfite treated DNA was amplified by PCR with BSP primers. PCR products were cloned into the pUC57 vector, and five clones selected and sequenced from each sample. Methylation level was defined as the ratio of methylated CpG sites in all clones. (A) Typical sequencing results; (B) early effects, tissues were collected 2 h postirradiation; (C) delay effects, 1 month postirradiation. *P,0.05 versus control.
Wang J, Zhang Y, Xu K, Mao X, Xue L, Liu X, Yu H, Chen L, Chu X. Genome-wide screen of DNA methylation changes induced by low dose X-ray radiation in mice. PLoS One. 2014 Mar 10;9(3):e90804.)
MeDIP-seq(甲基化DNA免疫共沉淀测序)
MeDIP-Seq(Methylated DNA Immunoprecipitation Sequencing)测序是基于抗体富集原理进行测序的全基因组甲基化检测技术,采用甲基化DNA免疫共沉淀技术,通过5'-甲基胞嘧啶抗体特异性富集 基因组上发生甲基化的DNA片段,然后通过高通量测序可以在全基因组水平上进行高精度的CpG密集的高甲基化区域研究。
技术流程
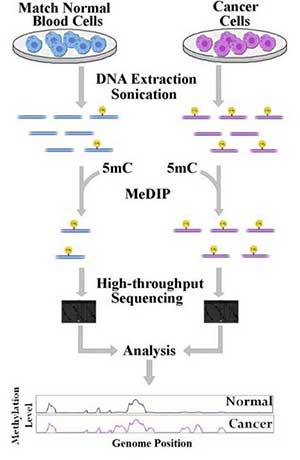
MeDIP流程图
技术运用
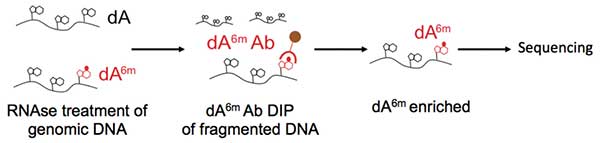
Illustration of dA6m DNA immunoprecipitation.
Genomic DNA is isolated and any RNAs are eliminated by RNAse treatment. Next, the DNA is fragmented and enriched by DNA immunoprecipitation using an antibody against dA6m. The enriched fraction can then be analyzed by subsequent deep sequencing.
(Koziol MJ, Bradshaw CR, Allen GE, Costa AS, Frezza C. Identification of Methylated Deoxyadenosines in Genomic DNA by dA(6m) DNA Immunoprecipitation. Bio Protoc. 2016 Nov 5;6(21).)
Distribution of genomic DNA fragmentation pattern after sonication.
5 μg of genomic DNA was fragmented in a volume of 300 μl for 35 min with the Diagenode sonicator. The size of the genomic DNA was determined by Tapestation analysis, with the D1000 Tapestation reagents. In this sample, the peak of the fragmented DNA was determined to be 245 bp. Lower and upper markers are also shown.
(Koziol MJ, Bradshaw CR, Allen GE, Costa AS, Frezza C. Identification of Methylated Deoxyadenosines in Genomic DNA by dA(6m) DNA Immunoprecipitation. Bio Protoc. 2016 Nov 5;6(21).)
三、转录后调控研究技术
RIP(RNA Binding Protein Immunoprecipitation)RNA结合蛋白免疫沉淀
RIP 技术(RNA Binding Protein Immunoprecipitation Assay,RNA 结合蛋白免疫沉淀)主要是运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA 进行q-PCR验证或者测序分析。RIP 是研究细胞内RNA 与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工具,可以帮助我们发现miRNA 的调节靶点。
根据客户需求,金开瑞可以提供RNA/蛋白、RNA/RNA互作验证RIP技术服务。
技术流程
载体构建及转染—细胞裂解液获取—磁珠的准备—RNA结合蛋白免疫沉淀—RNA纯化及鉴定(测序)
案例展示
1、使用目的蛋白寻找验证RNA(蛋白/RNA互作)
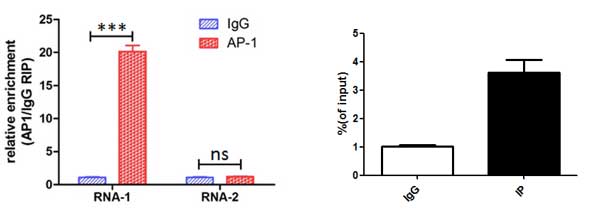
图. AP-1蛋白与RNA的相互作用RIP
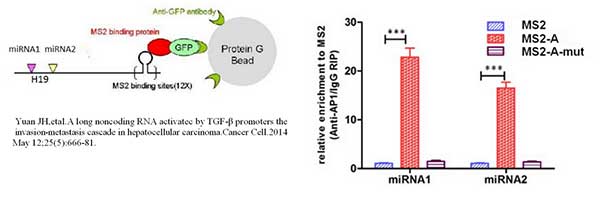
2、使用目的lncRNA寻找验证miRNA(RNA/RNA互作)
图. MS2- RIP实验原理图(表达Lnc-A的质粒富集miRNA1miRNA2的效率比不表达Lnc-A的MS2组明显提高。通过对照,说明MS2-A可以与miRNA1和miRNA2相互结合)
RNA pull down
使用体外转录法标记生物素RNA 探针,然后与胞浆蛋白提取液孵育,形成RNA-蛋白质复合物。该复合物可与链霉亲和素标记的磁珠结合,从而与孵育液中的其他成分分离。复合物洗脱后,通过western blot 实验检测特定的RNA 结合蛋白是否与RNA 相互作用。蛋白质与RNA 的相互作用是许多细胞功能的核心,如蛋白质合成、mRNA 组装、病毒复制、细胞发育调控等。若待检测目的蛋白明确,选择Western blot 鉴定;若不明确,则可选择质谱鉴定。
金开瑞现提供RNA pull down 检测技术服务, 使用特异性二抗进行检测,能有效避免抗体重链对结果的信号干扰。并且金开瑞配套有**上**的质谱仪,能显著提升检测效果。
技术流程
RNA 探针标记—胞浆蛋白提取与探针孵育—与磁珠结合、洗脱—WB检测或者质谱鉴定
案例展示
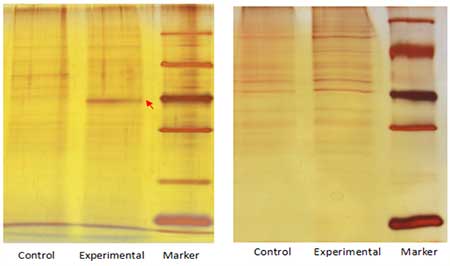
图. pull down下来的蛋白银染图。根据胶上条带的差异情况和实验目的,选择质谱或者WB鉴定pull down下来的蛋白

图.SDS-PAGE电泳后质谱鉴定
双荧光素酶报告系统
双荧光素酶报告基因用于实验系统中作相关的或成比例的检测,通常一个报告基因作为内对照,使另一个报告基因的检测均一化。检测基因表达时双报告基因通常用来瞬时转染培养细胞,带有实验报告基因的载体共转染带有不同的报告基因作为对照的第二个载体。通常实验报告基因偶联到调控的启动子,研究调控基因的结构和生理基础。报告基因表达活力的相对改变与偶联调控启动子转录活力的改变相关,偶联到组成型启动子的第二个报告基因,提供转录活力的内对照。
使测试不被实验条件变化所干扰。通过这种方法, 可减少内在的变化因素所削弱的实验准确性, 如培养细胞的数目和活力的差别, 细胞转染和裂解的效率。理想的双报告基因方法应该使用户能够以萤火虫荧光素酶所具有的速度,灵敏和线性范围对同一样品中的两个报告基因同时测定。金开瑞应用Promega 公司的双荧光素酶报告基因检测(DLR)系统,提供DLR 检测服务。甲基化特异性PCR(MSP技术)。
技术流程
序列分析—合成或者扩增目的片段亚克隆至目标载体—报告基因质粒、转录因子共转染细胞—荧光素酶反应发光—结果分析
案例展示
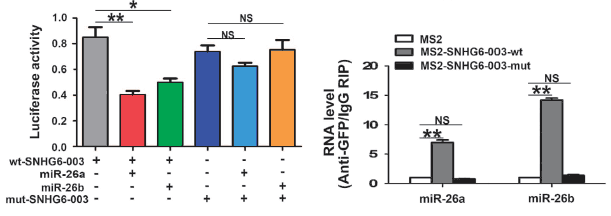
图. 双荧光素酶和RIP验证lncRNA可以与miR-26a结合
(C Cao, Zhang T, Zhang D, et al.The long non-coding RNA, SNHG6-003, functions as a competing endogenous RNA to promote the progression of hepatocellular carcinoma. Oncogene. 2017 Feb 23;36(8):1112-1122. )